
[DNA, Deoxy ribo Nucleic Acid]
デオキシリボ核酸。


RNAとともに核酸の一。
ある生物の全DNAをゲノムといい、ヒトでは99%が核内にあり、残りの1%はミトコンドリアにあるが、これらは原核細胞タイプのDNAであり、20億年前には独立して酸素呼吸をしていた原核生物シアノバクテリアが、真核細胞たる宿主に取り込まれたことを示す名残であると考えられている。
【構造】
◆縦列反覆(tandem repeat)非コード域に多いが、DNAの大部分
塩基対の種々の反覆(1回から数百回の反復)
このパターンによって制限酵素で切れる部分間の長さが変わる
(制限酵素断片長多型 RFLP: restriction fragment length polymorphism)
DNA指紋(DNA finger print)に利用
個体によってDNA構造は、大きく異なる。
【特徴】
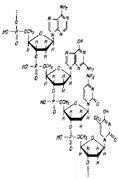
◆二重らせん構造
J.D.WatsonとF.H.C.Crickのモデル(1953)
(DNA塩基組成の化学研究・DNA結晶のX線解析)
・螺旋の直径は 2nm
糖・リン酸の基本骨格2本が外縁
・螺旋内部で隣り合う塩基対同士は、0.34nmの隔たり
・水素結合(プリンとピリミジンの間。5'末端-3'末端)
・塩基対を作る二つの塩基は同一平面状(螺旋軸に対して直交)
・36°ずつ回転(10塩基対/螺旋1回転; 1巻きで 3.4nm 進む)
◆環状
ほか、二本鎖、直鎖状などの形態
◆裸(ヒストンなどを持たない)
・RNAよりも不活性(安定)
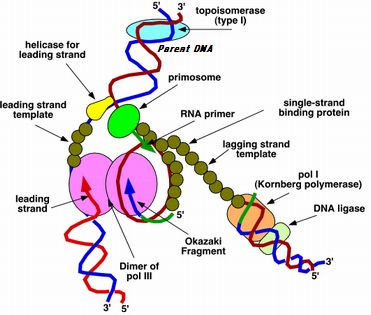
【DNAの複製】
新生鎖
5'末端から3'末端へ伸長してθ構造を形成(若干のウイルス・プラスミドでは逆方向)
・リーディング鎖
複製フォークの移動と同方向に重合が進むDNA鎖
・ラギング鎖(岡崎断片1,000〜2,000a.a.)
複製フォークの移動と逆方向に重合が進むDNA鎖
巻き取りと逆向きに合成
DNAリガーゼによって互いに連結される
原核生物では2つ以上ある複製起点から始まる
・複製フォーク(合成部位)
・DNAポリメラーゼ(Pol III)
ポリメラーゼ連鎖反応(重合反応)を触媒
プライマーを足がかりにDNAとの重合・合成(レプリソーム)
・プライマーRNA(プライモソーム)
ラギング鎖では周期的にプライマーゼによって作られる
・ヘリカーゼ(らせん不安定化タンパク質群)
巻き戻しを行う
・DNAジャイレース(クラスIIトポイソメラーゼ)
二本鎖の一方の鎖の切断と再結合
この際、適切な巻き戻しの程度の調整を行う
・SSBタンパク質(single strand-binding)
一本鎖をヌクレアーゼによる加水分解から保護
・DNAポリメラーゼ(Pol I エキソヌクレアーゼ活性による)
複製の終わったラギング鎖の下流のRNAプライマーを除去
ポリメラーゼ活性でDNAに置き換え、DNAリガーゼが岡崎断片を連結
【特徴】
◆機能
○遺伝子
大腸菌などの原核細胞では、DNAの殆どが遺伝子
真核細胞では遺伝子(エクソンexon)は、全DNAの3%以下
97%(junk DNA)は、イントロン(intron)と、未決定のDNA
○複製
・RNAから逆転写(RNAの遺伝情報を継承)
自己複製はRNAの助けを借りないとできない
・半保存的複製(有糸分裂)
大腸菌をN15標識して密度勾配遠心法で証明(Meselson&Stahl,1958)
その後、原核細胞・真核細胞のDNAに共通であることがわかった
・ヌクレオチドの重合速度は非常に速い
哺乳類で毎秒約50ヌクレオチド
細菌では毎秒約500ヌクレオチド
◆マーカー
propidium iodide
◆DNA多型(DNA polymorphism)
◆損傷
○原因
・脱プリン反応(プロトンによるアデニン、グアニンの切断)
・脱アミノ反応(水によるNH2からC=Oへの変化)
・チミン2量体生成(紫外線による損傷)
・アルキル化
・アルキル化剤(プリン塩基のN,O原子をアルキル化)
・S-アデノシルメチオニン(グアニンのN7をメチル化)
・活性酸素による酸化(OHラジカルがグアニンC8-HをC-OHに変化)
○結果
・転写活性化
・アポトーシス
・細胞周期チェックポイント
・DNA修復
・損傷寛容
【解析】
◆配列決定法
・サザン・ブロッティング
・DNAシークエンシング
・マイクロアレイ
・RFLP(restriction fragment length polymorphism)
制限酵素断片の比較
・RAPD法
10-mer程度のプライマーでPCRしてDNAのランダムな部位(<3000bp)を増幅
DNA濃度やtaq DNA polymeraseなどの条件でパターン再現性低い問題
・AP-PCR (arbitrary primed PCR)20-34mer
・DAMD (directed amplification of minisatellite region DNA)
・ISSR (Intersimple sequence repeat)
・STR(short tandem repeat) analysis
反復配列の長さの比較
◆音楽に変換
・Gene2Music
(Takahashi&Miller,2007; Takahashi,Klionsky,2012)
2013/05/22 masashi tanaka